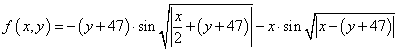Пример поиска минимума функцииАрхив проекта: ga_9.zip Продолжаем рассматривать примеры реализаций ГА и на этом занятии увидим, как с помощью эволюционного моделирования можно заставить популяцию устремляться в область минимума функции. Сейчас мы разберемся как это работает. Функции Eggholder и ХиммельблауПервым делом нам нужно выбрать функцию, у которой будем искать глобальный минимум. Благо, для этого уже существует определенный набор тестовых функций. Функция EggholderФункция Eggholder (ее форма напоминает подставку для яиц):
Обычно рассматривается на диапазоне [-512; 512] по каждой из координат и выглядит, следующим образом:
Особенностью этой функции является большое число локальных минимумов, где алгоритм поиска может легко застрять. Функция ХиммельблауВторая функция Химмельблау имеет математический вид:
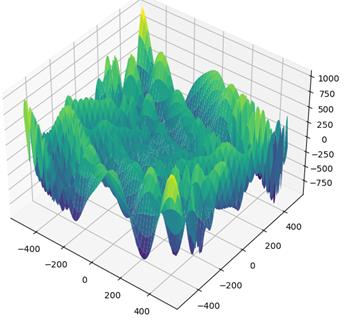
и следующее трехмерное отображение в пределах [-5;5] по осям x, y:
Особенность этой функции в том, что она имеет четыре глобальных оптимума в точках: (3.0; 2.0), (-2.805118; 3.131312), (-3.779310; -3.283186), (3.584458; -1.848126) Я выберу вторую функцию Химмельблау для поиска одного из глобальных минимумов с помощью ГА. Функции пакета DEAP для вещественных чиселТак как в генах хромосом мы будем хранить вещественные координаты точек (x; y), то нам потребуются функции для скрещивания и мутации, работающие с вещественными числами. Для этого в пакете DEAP предусмотрены следующие функции:
Я воспользуюсь двумя последними функциями (cxSimulatedBinaryBounded и mutPolynomialBounded) для выполнения операций скрещивания и мутаций, так как мы ограничим область поиска решений квадратом [-5; 5] по каждой координате x и y. Реализация алгоритма на PythonТеперь, когда мы определились с общей постановкой задачи, пришло время реализовать ее на Python с использованием пакета DEAP. Вначале, как всегда, сделаем импорт необходимых пакетов и модулей: from deap import base, algorithms from deap import creator from deap import tools import algelitism import random import matplotlib.pyplot as plt import numpy as np Затем, определим константы работы ГА, создадим объект для «зала славы» и установим «зерно» генератора случайных чисел, равное 42 (для повторяемости результатов): LOW, UP = -5, 5 ETA = 20 LENGTH_CHROM = 2 # длина хромосомы, подлежащей оптимизации # константы генетического алгоритма POPULATION_SIZE = 200 # количество индивидуумов в популяции P_CROSSOVER = 0.9 # вероятность скрещивания P_MUTATION = 0.2 # вероятность мутации индивидуума MAX_GENERATIONS = 50 # максимальное количество поколений HALL_OF_FAME_SIZE = 5 hof = tools.HallOfFame(HALL_OF_FAME_SIZE) RANDOM_SEED = 42 random.seed(RANDOM_SEED) Далее, объявляем два уже стандартных класса для представления индивидуума в пакете DEAP: creator.create("FitnessMin", base.Fitness, weights=(-1.0,)) creator.create("Individual", list, fitness=creator.FitnessMin) И, затем, нам нужна функция для генерации начальных значений генов в хромосомах нулевой популяции. Для этого воспользуемся функцией uniform – генерации СВ с равномерным законом в диапазоне [a; b]: def randomPoint(a, b): return [random.uniform(a, b), random.uniform(a, b)] Функция randomPoint возвращает список из двух случайных значений – начальные координаты (x; y) в хромосоме особи:
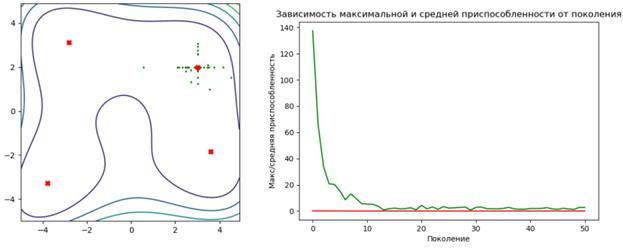
После этого, создаем ее алиас с именем randomPoint и параметрами a=LOW, b=UP (то есть, генерируем случайные значения в диапазоне [-5; 5]): toolbox = base.Toolbox() toolbox.register("randomPoint", randomPoint, LOW, UP) toolbox.register("individualCreator", tools.initIterate, creator.Individual, toolbox.randomPoint) toolbox.register("populationCreator", tools.initRepeat, list, toolbox.individualCreator) population = toolbox.populationCreator(n=POPULATION_SIZE) С помощью функции individualCreator создаем отдельную особь, а затем, функцией populationCreator – начальную популяцию. Далее, нам потребуется функция для вычисления приспособленности каждой особи. Здесь все просто. Используя координаты (x, y) в генах хромосомы, вычисляем функцию Химмельблау и возвращаем результат в виде кортежа: def himmelblau(individual): x, y = individual f = (x ** 2 + y - 11) ** 2 + (x + y ** 2 - 7) ** 2 return f, Осталось зарегистрировать необходимые функции для отбора, скрещивания, мутации и вычисления приспособленности: toolbox.register("evaluate", himmelblau) toolbox.register("select", tools.selTournament, tournsize=3) toolbox.register("mate", tools.cxSimulatedBinaryBounded, low=LOW, up=UP, eta=ETA) toolbox.register("mutate", tools.mutPolynomialBounded, low=LOW, up=UP, eta=ETA, indpb=1.0/LENGTH_CHROM) stats = tools.Statistics(lambda ind: ind.fitness.values) stats.register("min", np.min) stats.register("avg", np.mean) Здесь использованы стандартные функции cxSimulatedBinaryBounded() и mutPolynomialBounded() пакета DEAP для реализации скрещивания и мутации. А также, уже знакомая нам функция, selTournament() для турнирного отбора. По идее, уже можно запускать ГА, собирать статистику и наслаждаться результатом. Но мы добавим еще один элемент – интерактивное отображение множества решений на каждой итерации этого алгоритма. Вначале пропишем функцию show() для показа графика Химмельблау в виде линий уровня и популяции текущих решений в виде точек на нем: import time def show(ax, xgrid, ygrid, f): ptMins = [[3.0, 2.0], [-2.805118, 3.131312], [-3.779310, -3.283186], [3.584458, -1.848126]] ax.clear() ax.contour(xgrid, ygrid, f) ax.scatter(*zip(*ptMins), marker='X', color='red', zorder=1) ax.scatter(*zip(*population), color='green', s=2, zorder=0) plt.draw() plt.gcf().canvas.flush_events() time.sleep(0.2) Затем, создадим необходимые объекты для отображения этого графика: x = np.arange(-5, 5, 0.1) y = np.arange(-5, 5, 0.1) xgrid, ygrid = np.meshgrid(x, y) f_himmelbalu = (xgrid**2 + ygrid - 11)**2 + (xgrid + ygrid**2 - 7)**2 plt.ion() fig, ax = plt.subplots() fig.set_size_inches(5, 5) ax.set_xlim(LOW-3, UP+3) ax.set_ylim(LOW-3, UP+3) И запустим ГА: population, logbook = algelitism.eaSimpleElitism(population, toolbox, cxpb=P_CROSSOVER, mutpb=P_MUTATION, ngen=MAX_GENERATIONS, halloffame=hof, stats=stats, callback=(show, (ax, xgrid, ygrid, f_himmelbalu)), verbose=True) maxFitnessValues, meanFitnessValues = logbook.select("min", "avg") best = hof.items[0] print(best) plt.ioff() plt.show() Смотрите, здесь на каждой итерации будет вызываться функция show() с переданными ей параметрами. В результате, мы будем видеть, как изменяется популяция решений от поколения к поколению. Ну, а в самом конце, отобразим собранную статистику: plt.plot(maxFitnessValues, color='red') plt.plot(meanFitnessValues, color='green') plt.xlabel('Поколение') plt.ylabel('Макс/средняя приспособленность') plt.title('Зависимость максимальной и средней приспособленности от поколения') plt.show() После запуска программы увидим следующие графики:
При этом, лучшая хромосома содержит координаты точки: [2.999095667625665, 1.9999101547638773] что близко к оптимальному значению (3.0; 2.0). Вот так можно реализовывать ГА для вещественных значений в генах хромосом. Видео по теме |